近期,中国科学技术大学翁建平教授团队在Journal of Advanced Research杂志(IF=12.822)在线发表题为Machine learning approach reveals microbiome, metabolome, andlipidome profiles in type 1 diabetes[1]的研究论文。团队博士研究生谭慧玲、硕士研究生施雨、博士研究生岳彤为论文共同第一作者,通讯作者为翁建平教授和郑雪瑛副主任医师。该研究使用多种机器学习算法对1型糖尿病(T1D)的多组学特征进行了刻画,为进一步T1D防治靶点的发现提供了新思路。现分享其精粹,同君共阅!

多组学技术是刻画T1D异质性特征的良好手段
T1D作为一种免疫介导的慢性疾病,其发病机制涉及复杂的遗传、环境和免疫因素相互作用。通过整合基因组学、微生物组学以及代谢组学等多层次的数据,多组学技术能够全面、系统地揭示T1D患者之间的差异,从而深入了解疾病的发展过程和个体间的异质性。这些综合性的数据分析有助于识别潜在的生物标志物、理解疾病的亚型及其生物学特征,为个体化治疗和精准医学的实现提供了坚实的基础。因此,多组学技术在揭示T1D的异质性特征方面具有独特而强大的应用潜力。同时,该团队前期研究已经发现不同免疫表型的T1D患者多组学特征[2]、不同遗传背景的T1D患者多组学特征[3]均存在异质性。
基于研究背景和前期基础,该研究通过16S rRNA基因扩增子测序和液相色谱-质谱法分析肠道微生物群、血清代谢物和脂质,利用逻辑回归、支持向量机、高斯朴素贝叶斯和随机森林等机器学习方法进行综合分析。研究结果揭示了T1D患者的特征性微生物、代谢物和脂质,并进一步在验证组中得到了证实(图1展示了1型糖尿病患者的部分肠道菌群特征)。特征性微生物、代谢物和脂质的发现为T1D的早期精准诊断和未来防治靶点的筛选提供了坚实的前期基础和新视角。
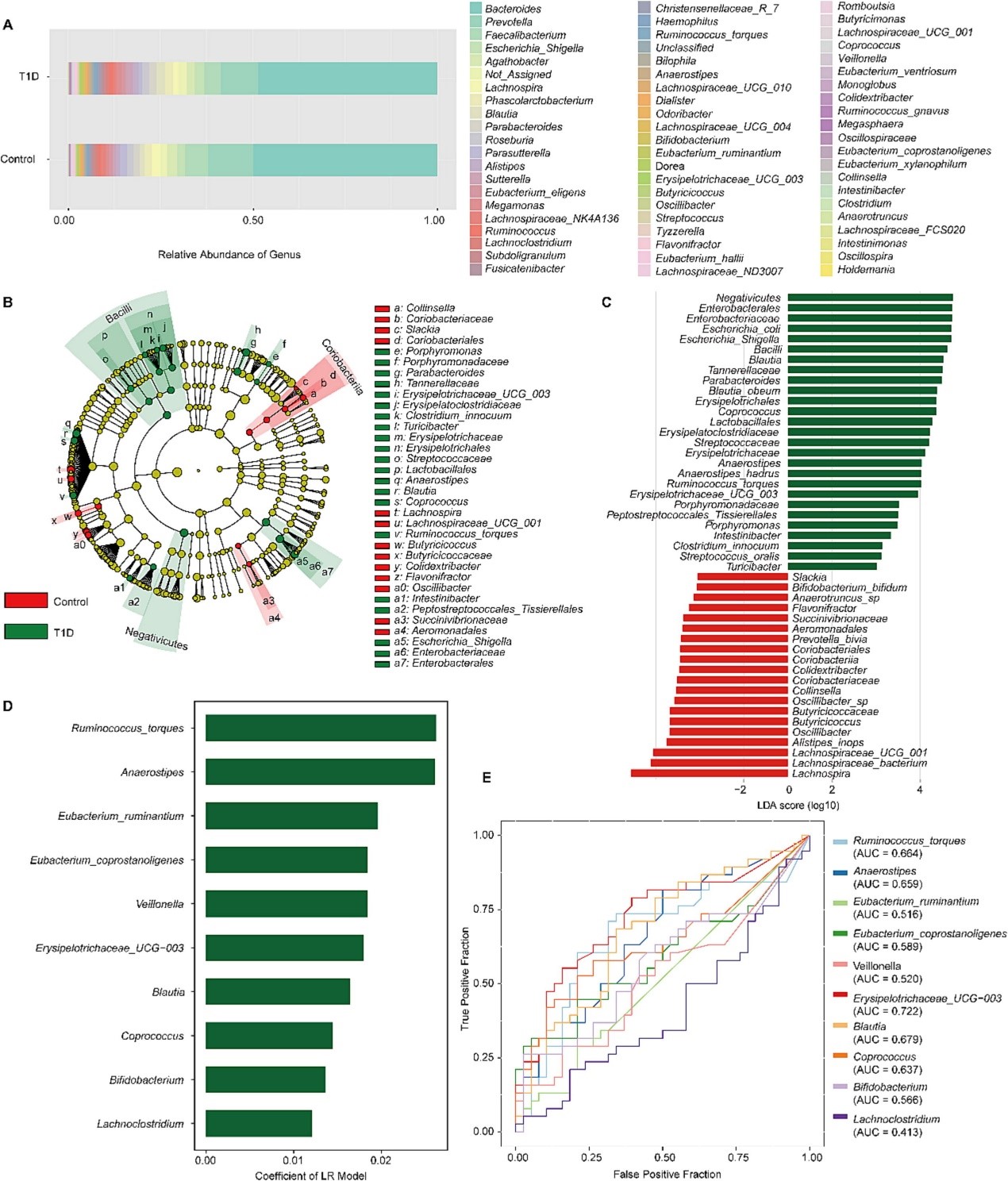
图1:1型糖尿病患者的肠道菌群特征
学科交叉:使用生信手段刻画T1D多组学特征
翁建平教授表示:该项研究是团队传统实验研究与现代技术结合的good paradigm,也是团队学科交叉尝试的第一个成果。生物信息学技术能够整合和分析大规模的多组学数据,更全面、深入地了解T1D患者的分子特征、生物学过程以及不同组学层次之间的相互关系。该研究采用机器学习和WGCNA(Weighted Gene Co-expression Network Analysis)等生物信息学分析方法对38例T1D患者和38例健康对照者的多组学特征进行分析,并使用61例T1D患者作为验证组。通过整合机器学习和WGCNA的结果,该研究发现代谢物3-羟基丁酸和9-氧代氧化物是T1D患者的特征血清代谢物(图2展示了1型糖尿病患者的部分血清代谢物特征)。可见,结合生物信息学、多组学、临床医学的多学科交叉分析为T1D的研究提供了强有力的工具,也为未来精准医学的发展奠定了一定的基础。
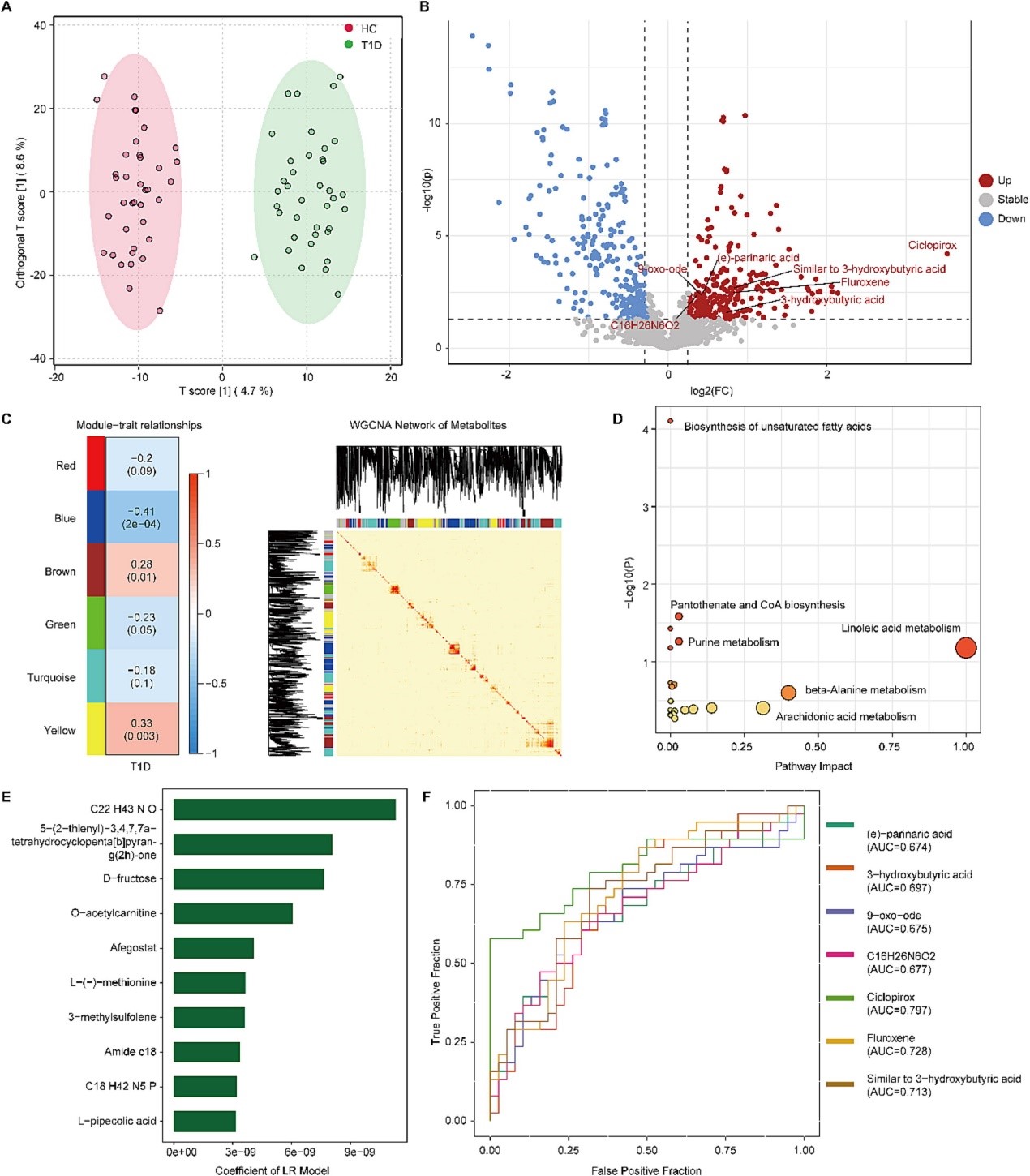
图2:1型糖尿病患者的血清代谢物特征
参考文献
[1] TAN H, SHI Y, YUE T, et al. Machine learningapproach reveals microbiome, metabolome, and lipidome profiles in type 1diabetes [J]. Journal of Advanced Research, 2023,
[2] LUO S, YUE T, LIU Z, et al. Gut microbiome and metabolic activityin type 1 diabetes: An analysis based on the presence of GADA [J]. FrontEndocrinol (Lausanne), 2022, 13(938358.
[3] YUE T, TAN H, WANG C, et al. High-risk genotypes for type 1diabetes are associated with the imbalance of gut microbiome and serummetabolites [J]. Front Immunol, 2022, 13(1033393.
